Page 170 - 《水产学报》2023年第1期
P. 170
刘杨,等 水产学报, 2023, 47(1): 019612
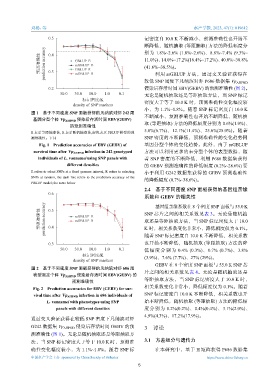
0.5 记密度自 10.0 K 不断减小,预测准确性也开始不
断降低,随机抽取 (等距抽取) 方法的降低幅度分
别为
1.8%~2.6% (1.8%~2.6%)、8.8%~7.4% (9.3%~
预测准确性 prediction accuracy 0.4 GBLUP_E 11.0%)、14.0%~17.2% (16.4%~17.2%)、40.0%~38.8%
ssGBLUP_E
(41.6%~38.5%)。
GBLUP_R
0.3
方法,通过交叉验证获得在
ssGBLUP_R
ssGBLUP
较低 利用 密度下凡纳滨对虾 P686 数据集 Vp AHPND
SNP
0.2 侵染后存活时间 EBV(GEBV) 的预测准确性 (图 2)。
50.0 30.0 10.0 1.0 0.1 无论是随机抽取还是等距抽取方法,当 SNP 标记
标记密度/K 密度大于等于 10.0 K 时,预测准确性变化幅度较
density of SNP markers
小,为 1.1%~1.8%。随着 SNP 标记密度自 10.0 K
图 1 基于不同密度 SNP 面板获得的凡纳滨对虾 242 尾
不断减小,预测准确性也开始不断降低,随机抽
基因分型个体 Vp AHPN 侵染后存活时间 EBV(GEBV)
D
取 (等距抽取) 方法的降低幅度分别为 0.6%(1.9%)、
的预测准确性
5.8%(6.7%)、12.1%(11.4%)、25.6%(25.0%)。随着
E 表示等距抽取法, R 表示随机抽取法,虚线表示 PBLUP 模型的预
测准确性;下同 SNP 密度的不断降低,预测准确性的变化趋势同
Fig. 1 Prediction accuracies of EBV (GEBV) of 基因分型个体的变化趋势。此外,由于 ssGBLUP
survival time after Vp AHPND infection in 242 genotyped 方法可以利用更多的未分型个体的表型数据,随
individuals of L. vannamei using SNP panels with 着 SNP 密度的不断降低,利用 P686 数据集获得
different densities 的 GEBV 预测准确性的降低幅度 (0.2%~28.6%) 要
E refers to select SNPs at a fixed genome interval, R refers to selecting 小于利用 G242 数据集获得的 GEBV 预测准确性
SNPs at random, the dash line refers to the prediction accuracy of the
的降低幅度 (0.7%~38.6%)。
PBLUP model; the same below
2.4 基于不同密度 SNP 面板获得的基因组亲缘
0.6
系数和 基因组亲缘系数在 8 个密度 SNP 面板与 55.0 K
的相关性
GEBV
预测准确性 prediction accuracy 0.5 ssGBLUP_E SNP 时,相关系数变化非常小,降低幅度仅为 0.1%。
芯片之间的相关系数见表
3。无论是随机抽
取还是等距抽取方法,当
标记密度大于
10.0
SNP
ssGBLUP_R
0.4
K
也开始不断降低,随机抽取
(等距抽取) 方法的降
0.3 随着 SNP 标记密度自 10.0 K 不断降低,相关系数
50.0 30.0 10.0 1.0 0.1 低 幅 度 分 别 为 0.4% (0.3%)、 0.7% (0.7%)、 3.8%
标记密度/K (3.9%)、7.6% (7.7%)、27% (29%)。
density of SNP markers
GEBV 在 8 个密度 SNP 面板与 55.0 K SNP 芯
图 2 基于不同密度 SNP 面板获得的凡纳滨对虾 686 尾
片之间的相关系数见表 4。无论是随机抽取还是
表型测定个体 Vp AHPN 侵染后存活时间 EBV(GEBV) 的
D
等距抽取方法,当 SNP 标记密度大于 10.0 K 时,
预测准确性
相关系数变化非常小,降低幅度仅为 0.1%。随着
Fig. 2 Prediction accuracies for EBV (GEBV) for sur-
vival time after Vp AHPND infection in 686 individuals of SNP 标记密度自 10.0 K 不断降低,相关系数也开
L. vannamei with phenotypes using SNP 始不断降低,随机抽取 (等距抽取) 方法的降低幅
panels with different densities 度分别为 0.2%(0.2%)、0.4%(0.4%)、3.1%(2.0%)、
4.9%(4.3%)、17.2%(17.9%)。
通过交叉验证获得在较低 SNP 密度下凡纳滨对虾
G242 数据集 Vp AHPN D 侵染后存活时间 GEBV 的预 3 讨论
测准确性 (图 1)。无论是随机抽取还是等距抽取方
法,当 SNP 标记密度大于等于 10.0 K 时,预测准 3.1 方差组分与遗传力
确性变化幅度较小,为 1.1%~1.8%。随着 SNP 标 在本研究中,基于 H 矩阵获得 P686 数据集
中国水产学会主办 sponsored by China Society of Fisheries https://www.china-fishery.cn
5