Page 171 - 《水产学报》2023年第1期
P. 171
刘杨,等 水产学报, 2023, 47(1): 019612
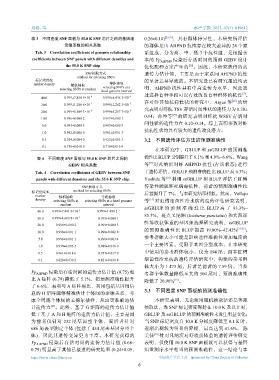
表 3 不同密度 SNP 面板与 55.0 K SNP 芯片之间的基因组 0.26±0.10) [25-27] ,具有群体特异性。本研究所评估
亲缘系数的相关系数 的群体是由 AHPND 抗性存在较大差异的 24 个家
Tab. 3 Correlation coefficients of genomic relationship 系组成,分为高、中、低 3 个抗性组,是根据亲
coefficients between SNP panels with different densities and 本的 Vp AHPN D 侵染后存活时间的预测 GEBV 设计
the 55.0 K SNP chip 优化配种方案产生的 。因此,本研究获得的高
[28]
SNP抽取方式 遗传力估计值,主要是由于家系间 AHPND 抗性
method for selecting SNPs
标记密度/K 的显著差异导致的。本研究及已有研究报道均表
marker density 随机抽取 等距抽取
selecting SNPs at random selecting SNPs at a 明,AHPND 抗性具有中高遗传力水平,因此通
fixed genome interval
[7]
过选择育种手段可以有效改良育种群体的抗性 。
40.0 0.999±7.858 9×10 −6 0.999±6.436 1×10 −6
在对虾其他抗病性状的研究中,Argue 等 [26] 的研
30.0 0.999±1.286 6×10 −5 0.999±2.262 3×10 −5
究表明对虾抗 TSV 存活时间性状的遗传力为 0.28±
20.0 0.999±5.509 3×10 −5 0.999±5.257 7×10 −5
0.14;孙坤等 [29] 的研究表明对虾抗 WSSV 存活时
10.0 0.996±0.000 2 0.997±0.000 1
间性状的遗传力在 0.10~0.14。综上表明多数对虾
5.0 0.993±0.000 3 0.993±0.000 3
抗病性状均具有较大的遗传改良潜力。
1.0 0.962±0.001 6 0.961±0.001 3
0.5 0.924±0.004 0 0.923±0.003 3 3.2 不同遗传评估方法的预测准确性
0.1 0.730±0.018 0 0.710±0.016 9
在本研究中,GBLUP 和 ssGBLUP 的预测准
表 4 不同密度 SNP 面板与 55.0 K SNP 芯片之间的 确性比 BLUP 分别提升了 6.1% 和 4.9%~6.6%。Wang
[7]
GEBV 相关系数 等 对凡纳滨对虾 AHPND 抗性 (存活状态) 进行
Tab. 4 Correlation coefficients of GEBV between SNP 了遗传评估,GBLUP 预测准确性比 BLUP 高 6.3%;
panels with different densities and the 55.0 K SNP chip Yoshida 等 [30] 利用 ssGBLUP 和 BLUP 评估了虹鳟
SNP抽取方式 传染性胰脏坏死病毒抗性,前者的预测准确性比
标记密度/K method for selecting SNPs 后者提升了
marker 随机抽取 等距抽取 7%,与本研究结果相似。然而,Vallejo
density [15]
selecting SNPs at selecting SNPs at a fixed genome 等 对虹鳟细菌性冷水病的遗传评估研究表明,
random interval
ssGBLUP 的 预 测 准 确 性 比 BLUP 高 了 83.3%~
40.0 0.999±7.441 0×10 −6 0.999±1.490 2 −5
85.3%。斑点叉尾鮰 (Ictalurus punctatus) 和大西洋
30.0 0.999±4.603 8×10 −5 0.999±0.000 1
鲑等收获体重的基因组选择研究表明,ssGBLUP
20.0 0.999±0.000 2 0.999±0.000 3
的预测准确性比 BLUP 提高 19.00%~42.42% [31-32] 。
10.0 0.998±0.001 1 0.998±0.002 8
参考群体大小可能是影响选择准确性增加幅度的
5.0 0.996±0.001 1 0.996±0.003 4
一个主要因素。受限于基因分型成本,在本研究
1.0 0.979±0.006 5 0.980±0.016 0
中使用的参考群体较小,仅为 尾;而在虹鳟
0.5 0.961±0.014 6 0.957±0.017 0 194
0.1 0.828±0.030 2 0.821±0.038 8 细菌性冷水病的遗传评估研究中,构建的参考群
体大小为 1 473 尾,后者是前者的 7.59 倍。当参
Vp AHPN D 侵染后存活时间的遗传力估计值 (0.75) 相 考群个体数量降低至大约 500 尾时,预测准确性
比 A 矩阵 (0.79) 降低了 5.1%,但预测准确性提升 降低了 20.90% 。
[15]
了 6.6%,表明与 A 矩阵相比,利用包括基因组信
3.3 不同密度 SNP 面板的预测准确性
息的 H 矩阵能够精准估计个体间的亲缘关系,考
虑全同胞个体间的孟德尔抽样,从而更准确地估 本研究表明,无论使用随机抽取法还是等距
[24]
计遗传力 。此外,基于 G 矩阵的遗传力估计值 抽取法,当 SNP 标记密度保持在 10.0 K 及以上时,
低于基于 A 和 H 矩阵的遗传力估计值,主要是因 GBLUP 及 ssGBLUP 的预测准确性未发生明显变化;
为前者仅针对 232 尾基因型个体,而后者针对 当 SNP 标记密度自 10.0 K 分梯度降低至 0.1 K 时,
686 尾表型测定个体 (包括了 454 尾未基因分型个 逐渐出现较为明显的降幅,最高达到 41.6%。 陈
体),因此其遗传变异更为丰富。本研究获得的 美佳 [16] 针对凡纳滨对虾收获体重的遗传评估研究
Vp D 侵染后存活时间的遗传力估计值 (0.68~ 表明,仅使用 10.0 K SNP 面板就可以获得与基因
AHPN
0.79) 明显高于其他已报道的研究结果 (0.24±0.09, 组重测序水平相当的预测准确性,这一结论与本
https://www.china-fishery.cn 中国水产学会主办 sponsored by China Society of Fisheries
6