Page 223 - 《软件学报》2021年第5期
P. 223
宋杰 等:基于深度学习的数字病理图像分割综述与展望 1447
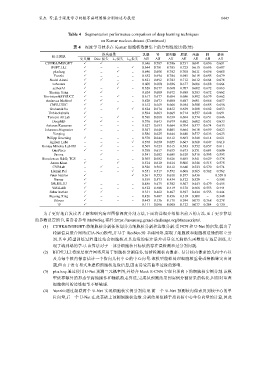
Table 4 Segmentation performance comparison of deep learning techniques
on Kumar nucleus dataset (Continued)
表 4 深度学习技术在 Kumar 细胞核数据集上的分割性能比较(续)
损失函数 乳腺 肾 前列腺 膀胱 结肠 肺 整体
相关团队
交叉熵 Dice 损失 L 1 损失 L 2 损失 AJI AJI AJI AJI AJI AJI AJI
CUHK&IMSIGHT 3 3 − − 0.646 0.707 0.706 0.731 0.641 0.696 0.691
BUPT.J.LI 3 − − 3 0.644 0.701 0.705 0.723 0.615 0.696 0.687
pku.hzq 3 − 3 − 0.646 0.698 0.702 0.708 0.611 0.696 0.685
Yunzhi 3 − − − 0.632 0.694 0.704 0.695 0.619 0.695 0.679
Navid Alemi 3 3 − − 0.621 0.692 0.703 0.712 0.612 0.684 0.678
xuhuaren 3 − 3 − 0.608 0.680 0.696 0.677 0.606 0.688 0.664
aetherAI 3 − 3 − 0.628 0.677 0.660 0.707 0.602 0.672 0.663
Shuang Yang 3 3 − − 0.624 0.680 0.672 0.688 0.583 0.672 0.662
Bio-totem&SYSUCC 3 − 3 − 0.617 0.677 0.684 0.686 0.592 0.679 0.662
Amirreza Mahbod 3 3 − − 0.628 0.673 0.680 0.687 0.601 0.666 0.657
CMU-UIUC 3 3 − − 0.612 0.669 0.666 0.694 0.580 0.685 0.656
Graham&Vu 3 − 3 3 0.624 0.674 0.652 0.639 0.580 0.682 0.653
Unblockabulls 3 − − − 0.594 0.663 0.665 0.714 0.557 0.648 0.651
Tencent AI Lab 3 − 3 − 0.596 0.668 0.659 0.684 0.574 0.651 0.646
DeepMD 3 3 − − 0.576 0.643 0.679 0.682 0.602 0.651 0.633
Antanas Kascenas 3 − − − 0.627 0.653 0.664 0.704 0.577 0.674 0.633
Johannes Stegmaier 3 − − − 0.547 0.646 0.665 0.664 0.610 0.659 0.623
Yanping − − − − 0.586 0.625 0.644 0.680 0.577 0.633 0.623
Philipp Gruening 3 − − − 0.570 0.644 0.612 0.683 0.540 0.614 0.621
Agilent Labs − − − 3 0.570 0.650 0.655 0.667 0.560 0.647 0.618
Konica Minolta Lab EU − 3 − − 0.569 0.623 0.615 0.583 0.552 0.657 0.611
OnePiece 3 − − − 0.538 0.617 0.635 0.673 0.551 0.604 0.606
Junma − 3 − − 0.541 0.602 0.601 0.629 0.516 0.595 0.593
Biosciences R&D, TCS − − − − 0.565 0.602 0.626 0.685 0.541 0.629 0.578
Azam Khan 3 − 3 − 0.514 0.610 0.614 0.606 0.546 0.515 0.575
CVBLab − 3 − − 0.526 0.562 0.612 0.640 0.523 0.570 0.574
Linmin Pei − − − 3 0.551 0.517 0.592 0.608 0.565 0.582 0.562
Peter Naylor 3 − − 3 0.561 0.553 0.628 0.597 0.436 − 0.559 8
Kumar 3 − − − 0.539 0.573 0.434 0.522 0.529 − 0.508
DB-KR-JU 3 − − − 0.486 0.475 0.302 0.507 0.443 0.479 0.455
VISILAB 3 − − − 0.422 0.406 0.419 0.378 0.468 0.553 0.444
Sabarinathan 3 3 − − 0.331 0.462 0.467 0.397 0.424 0.553 0.444
Fuyong Xing 3 − − − 0.426 0.407 0.456 0.319 0.308 − 0.348
Silvers 3 − − − 0.445 0.136 0.171 0.394 0.073 0.368 0.278
TJ 3 − − − 0.111 0.096 0.089 0.122 0.077 0.284 0.130
为了更好地启发读者了解和研究病理图像深度分割方法,下面将详细介绍排名前五的方法.至于更多算法
的参数设置细节,请读者参考 MoNuSeg 稿件:https://monuseg.grand-challenge.org/Manuscripts/.
(1) CUHK&IMSIGHT:将细胞核分割任务划分为细胞核分割和边缘分割.受 FCN 和 U-Net 的启发,提出了
轮廓信息聚合网络(CIA-Net)模型,并基于 ResNet-50 基础网络,实现了细胞核和细胞核边缘的联立分
割.其中,模型训练过程通过结合细胞核及其边缘的标注误差引导交叉熵损失函数进行逐层训练,实
现了端到端的学习.该算法对于一部分细胞核目标依然存在误检测和过分割问题.
(2) BUPT.J.LI:将深层聚合网络应用于细胞核分割任务,包括检测核内像素、估计核内像素的几何中心以
及为每个核内像素估计一个指向几何中心的中心向量.该模型能够处理细胞核重叠或接触等突出问
题,但由于没有显式地建模细胞核边缘信息,因而易受高曲率边缘的影响.
(3) pku.hzq:通过使用 U-Net 预测三元概率图,并结合 Mask R-CNN 实现自顶向下的细胞核实例分割.该模
型依靠额外的形态学腐蚀操作和随机游走算法,完成从预测结果到实例分割结果的转化,因而对重叠
细胞核间的边缘细节不够敏感.
(4) Yunzhi:通过级联两个 U-Net 实现细胞核实例分割结果.前一个 U-Net 预测核内像素及到核中心的单
位向量,后一个 U-Net 在此基础上预测细胞核边缘.分割结果依赖于指向核中心单位向量的计算,因此