Page 221 - 《软件学报》2021年第5期
P. 221
宋杰 等:基于深度学习的数字病理图像分割综述与展望 1445
()
*
其中,分割得到的连通成分 Si 利用 G i 得到 JI 最大值.
k
6 深度学习分割模型性能分析
本节首先在目前比较流行的分割基准公共数据集上对最新的深度学习算法的性能进行统计分析,然后,在
此基础上对不同类型的数字病理图像的分割性能进行评估和总结.需要注意的是:虽然大多数已有模型基于标
准分割指标均得到了综合性能评测,但是依然有一些模型未能做到,因此难以对它们全面比较.
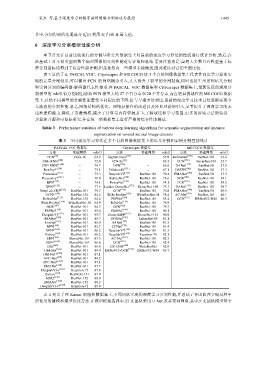
表 3 总结了在 PASCAL VOC、Cityescapes 和 MS COCO 这 3 个自然图像数据集上代表性深度学习算法实
现的定量分割结果.可以看出:FCN 的网络概念引入,大大提升了模型的分割精度,同时也衍生出系列语义分割
和实例分割的编码器-解码器代表性体系.在 PASCAL VOC 数据集和 Cityescapes 数据集上,最新发展的深度分
割模型的 mIoU 值分别超出原始 FCN 模型大约 27 个百分点和 20 个百分点,而在更具挑战性的 MS COCO 数据
集上,虽然不同模型的分割性能遭受不同程度的下降,但与早期方法相比,最新的深度学习技术已经逐渐实现令
人满意的分割性能.总之,网络结构的优化、网络层操作的改进以及经典理论的引入,显著提升了深度学习的表
达和建模能力,降低了参数规模,减少了计算与内存资源,扩大了跨域迁移学习范围.更多的后续方法期待进一
步算法开源和可复原研究,并在统一的数据集上进行严格的综合性能测试.
Table 3 Performance statistics of various deep learning algorithms for semantic segmentation and instance
segmentation on several natural image datasets
表 3 不同深度学习算法在多个自然图像数据集上的语义分割和实例分割性能统计
PASCAL VOC 数据集 Cityescapes 数据集 MS COCO 数据集
方法 基础网络 mIoU 方法 基础网络 mIoU 方法 基础网络 mIoU
FCN [16] VGG-16 62.2 SegNet basic [59] − 57.0 RefineNet [155] ResNet-101 33.6
CRF-RNN [140] − 72.0 FCN-8s [16] − 65.3 CCN [181] DenseNet-101 35.7
CRF-RNN* [140] − 74.7 DPN [152] − 66.8 DANet [177] ResNet-50 37.9
BoxSup* [150] − 75.1 Dilation10 [171] − 67.1 DSSPN [182] ResNet-101 37.3
Piecewise [151] − 75.3 DeeplabV2 [153] ResNet-101 70.4 EMANet [168] ResNet-50 37.5
Piecewise* [151] − 78.0 RefineNet [155] ResNet-101 73.6 SGR [183] ResNet-101 39.1
DPN [152] − 74.1 FoveaNet [172] ResNet-101 74.1 OCR [157] ResNet-101 39.5
DPN* [152] − 77.5 Ladder DenseNet [173] DenseNet-169 73.7 DANet [177] ResNet-101 39.7
DeepLab-CRF [153] ResNet-101 79.7 GCN [154] ResNet-101 76.9 EMANet [168] ResNet-50 39.9
GCN* [154] ResNet-152 82.2 Wide ResNet [156] WideResNet-38 78.4 AC-Net [179] ResNet-101 40.1
RefineNet [155] ResNet-152 84.2 PSPNet [158] ResNet-101 85.4 OCR [157] HRNetV2-W48 40.5
Wide ResNet [156] WideResNet-38 84.9 BiSeNet [174] ResNet-101 78.9
OCR [157] ResNet-101 84.3 DFN [162] ResNet-101 79.3
PSPNet [158] ResNet-101 85.4 PSANet [160] ResNet-101 80.1
DeeplabV3 [159] ResNet-101 85.7 DenseASPP [175] DenseNet-161 80.6
PSANet [160] ResNet-101 85.7 SPGNet [176] 2xResNet-50 81.1
EncNet [161] ResNet-101 85.9 DANet [177] ResNet-101 81.5
DFN [162] ResNet-101 82.7 CCNet [178] ResNet-101 81.4
DFN* [162] ResNet-101 86.2 DeeplabV3 [159] ResNet-101 81.3
Exfuse [163] ResNet-101 86.2 DeeplabV3 [169] Xception-71 82.1
SDN [164] DenseNet-161 83.5 AC-Net [179] ResNet-101 82.3
SDN* [164] DenseNet-161 86.6 OCR [157] ResNet-101 82.4
DIS [165] ResNet-101 86.8 GS-CNN [180] WideResNet 82.8
DM-Net [166] ResNet-101 84.4 HRNetV2+OCR [157] HRNetV2-W48 83.7
DM-Net* [166] ResNet-101 87.1
APC-Net [167] ResNet-101 84.2
APC-Net* [167] ResNet-101 87.1
EMANet [168] ResNet-101 87.7
DeeplabV3+ [169] Xception-71 87.8
Exfuse [163] ResNeXt-131 87.9
MSCI [170] ResNet-152 88.0
EMANet [168] ResNet-152 88.2
DeeplabV3+* [169] Xception-71 89.0
表 4 列出了在 Kumar 细胞核数据集上,不同团队实现的深度学习分割性能,并总结了各团队在实现过程中
所使用的建模和模型优化方法.在模型框架选择中,22 支团队采用 U-Net 及其变种网络,其中,9 支团队模型基于